argparse: un outil méconnu
Utiliser le paquetage argparse pour faciliter l'appel de scripts R.

Avez-vous déjà eu à lancer un programme avec différents paramètres? J’imagine que oui … Une manière de faire serait de se définir des paramètres en début de programme, les changer manuellement, sauvegarder le programme et relancer de nouveau. Vous imaginez bien que cela n’est pas agréable si on veut tester 20 combinaisons de paramètres différents. Une autre manière pourrait être de se définir un fichier de configurations, mais encore là on se retrouve face au même problème de devoir définir plusieurs fichiers de configurations ou bien d’avoir un programme qui itère sur les configurations du fichier. Bon, on s’approche de quelque chose qui devient de moins en moins laborieux. Pour ma part, j’aime bien l’idée d’avoir un programme paramétrable et définir les différents appels dans un autre fichier de lancement.
Dans les derniers mois, j’ai été confronté à 2 défis qui m’ont éventuellement permis de découvrir ce concept
de paramétriser un programme, et ce, via un paquetage R. Dans un premier temps, je devais intégrer l’appel
de programmes R à l’intérieur d’un fichier de ligne de commande ou un shell script (un fichier avec
l’extension .sh) dans lequel je devais passer différents arguments qui allaient être utilisés
par le programme. Dans un deuxième temps, je devais lancer un programme R, avec différents paramètres, à
l’aide d’une instance en ligne (par exemple AWS), pour laquelle je n’avais pas d’interface
graphique. Dans les deux cas, je devais faire l’appel d’un programme R à partir de la ligne de commande, ce
qui est d’ailleurs possible avec la commande Rscript. Par contre, je devais également passer
des arguments à ce programme lors de l’appel à la ligne de commande. C’est à ce moment que j’ai fais la
découverte du paquetage argparse,
un outil méconnu par plusieurs et qui offre des fonctionnalités intéressantes.
argparse et la ligne de commande
Comme vous l’avez probablement deviné dans l’introduction, argparse est un paquetage R qui
permet de faciliter l’appel de programmes R à partir de la ligne de commande. Pour être plus précis,
argparse permet de créer une interface de ligne de commande (command line interface
ou CLI). Cette interface agit comme une sorte de pont (ou moyen de communication) entre l’appel
d’un programme via la ligne de commande et les opérations effectuées à l’intérieur de ce programme.
Pourquoi appeler un programme R de la ligne de commande alors qu’on peut le lancer directement de la majorité des IDE comme RStudio ou même à partir de R directement? Plusieurs exemples pourraient être cités, les deux mentionnés en introduction sont pour moi des applications courantes, surtout lorsqu’on doit utiliser des ressources en ligne comme des instances AWS pour plus de puissance de calculs. Ces instances sont souvent dépourvues d’interface graphique, ce qui fait en sorte que nous nous retrouvons souvent devant le terrifiant écran noir du terminal.
Fonctionnement
Le paquetage argparse est en fait un wrapper à la librairie Python du même nom.
D’autres paquetages R, comme
optparse, fonctionnent de
manière similaire. L’objectif de cet article n’est pas de vanter un paquetage plus qu’un autre, mais
plutôt d’illustrer le concept général en utilisant comme exemple le paquetage argparse. La
création d’une interface de ligne de commande avec ce paquetage est très simple, les étapes sont
généralement :
- Définir les arguments pouvant être appelés de la ligne de commande
- Parser les arguments et les stocker dans un objet (habituellement une liste)
- Utiliser les éléments de cette liste dans le programme
Les étapes 2 et 3 sont sont assez simples à réaliser. L’étape la plus cruciale est de bien définir les arguments (étape 1). Pour se faire, il y a quelques éléments essentiels à prendre en compte. Les prochaines sections mettent en lumière ces considérations.
Initialiser l’objet Parser
Tout d’abord, avant même de définir les arguments, il faut attacher le paquetage
argparse et initialiser l’objet de type Parser. Cet objet est le coeur de
l’interface entre la ligne de commande et le programme et permettra de définir et stocker des arguments.
Une fois cet objet initialisé, nous pourrons ajouter des arguments à celui-ci, ce que nous verrons dans
les prochaines sections.
library(argparse)
parser <- ArgumentParser(description = "Simuler des distributions normales")Nommer les arguments
Maintenant que l’objet Parser est défini, il faut définir et nommer les arguments qui
pourront être appelés à la ligne de commande. Pour ajouter ces arguments à notre objet, on utilise la
méthode add_argument(). Il existe deux sortes d’arguments: les argument positionnels et les
arguments optionnels. Dans le premier cas, ces arguments sont obligatoires et doivent être appelés dans
un ordre précis alors que dans le deuxième cas, ils sont facultatifs et peuvent être appelés dans
n’importe quel ordre, tant qu’ils sont nommés dans l’appel. Voici un exemple simple d’ajout de ces 2
sortes d’arguments:
parser$add_argument("n_dist", type = "integer")
parser$add_argument("-m", "--mean", type = "double", default = 0)
Les arguments optionnels sont généralement identifiés par le préfixe - alors que les autres
seront considérés comme positionnels. L’option required permet également de spécifier les
arguments qui ne peuvent pas être omis lors de l’appel.
Il est possible de spécifier le type de valeur de l’argument grâce à l’option type qui peut
prendre les types suivants:
- “double”
- “character”
- “logical”
- “integer”
Les arguments optionnels doivent être accompagnés d’une valeur par défaut. Notez que ceux-ci sont
parfois nommés de 2 manières, avec un seul trait d’union (-m) pour l’abréviation courte et
deux (--mean) pour le nom complet. Il n’est pas obligatoire de mettre les deux, mais cela
peut améliorer la clarté de l’appel.
Définir l’aide
Une fonctionnalité intéressante avec les interfaces de ligne de commande comme celle du paquetage
argparse est que l’on peut généralement définir une aide pour l’appel de chaque argument
pouvant être appelé par le programme. Cela permet de documenter un programme en résumant son objectif et
en résumant son mode d’usage. Cette aide peut être affichée en appelant l’argument -h ou
--help au programme. Par exemple,
parser <- ArgumentParser(description = "Simuler des distributions normales")
parser$add_argument("n_dist", type = "integer",
help = "nombre de distributions simulées")
parser$add_argument("-m", "--mean", type = "double", default = 0,
help = "moyenne pour chaque distribution normale [défault: %(default)s, type: %(type)s]")
parser$print_help()## usage: /Library/Frameworks/R.framework/Versions/3.6/Resources/library/blogdown/scripts/render_page.R
## [-h] [-m MEAN] n_dist
##
## Simuler des distributions normales
##
## positional arguments:
## n_dist nombre de distributions simulées
##
## optional arguments:
## -h, --help show this help message and exit
## -m MEAN, --mean MEAN moyenne pour chaque distribution normale [défault: 0,
## type: float]Notez que la valeur par défaut et le type peuvent être incorporés dans l’aide (voir le code ci-dessus).
Fonctionnalités supplémentaires
Ces quelques options devraient vous permettre de créer une interface de ligne de commande simple d’utilisation et couvrant la majorité de vos besoins. Toutefois, notez que le paquetage possède également plusieurs autres fonctionnalités intéressantes comme regrouper des arguments, hériter des propriétés d’autres arguments parents, améliorer l’affichage de l’aide, etc.
Une fois les arguments définis
Une fois les arguments bien définis, il ne reste plus qu’à parser les arguments passés à ligne
de commande et les utiliser dans le programme en utilisant la fonction parse_args():
args <- parser$parse_args()Exemple
Voici un exemple de programme où on veut simuler un certain nombre de lois normales et estimer la moyenne et l’écart-type de la distribution avec ces simulations. Supposons en plus qu’on veuille pouvoir spécifier les paramètres de ces lois normales et sauvegarder un graphique illustrant la densité estimée comparativement à la vraie densité.
library(argparse)
library(ggplot2)
# Batir l’interface de ligne de commande ----------------------------------
parser <- ArgumentParser(description = "Simuler des distributions normales")
parser$add_argument("n_dist", type = "integer",
help = "nombre de distributions à simuler")
parser$add_argument("-m", "--mean", type = "double", default = 0, metavar = "",
help = "moyenne pour chaque distribution normale [défault: %(default)s, type: %(type)s]")
parser$add_argument("-s", "--sd", type = "double", default = 1, metavar = "",
help = "écart-type pour chaque distribution normale [défault: %(default)s, type: %(type)s]")
parser$add_argument("-n", "--n-obs", type = "integer", default = 1000, metavar = "",
help = "nombre d'observations simulés dans chaque simulation [défault: %(default)s, type: %(type)s]")
parser$add_argument("-r", "--random-seed", type = "integer", default = 42, metavar = "",
help = "nombre aléatoire d'amorce [défault: %(default)s, type: %(type)s]")
parser$add_argument("-g", "--graph-save", action = "store_true",
help = "sauvegarder le graphique [défault: %(default)s]")
parser$add_argument("-p", "--path-graph", type = "character", default = "./graph.png", metavar = "",
help = "chemin vers lequel sauvegarder le graphique [défault: \"%(default)s\"]")
args <- parser$parse_args()
# Simuler les lois normales -----------------------------------------------
set.seed(args$random_seed)
simulated_list <- lapply(1:args$n_dist, function(x) sort(rnorm(n = args$n_obs, mean = args$mean, sd = args$sd)))
simulated_matrix <- matrix(unlist(simulated_list), nrow = args$n_dist, ncol = args$n_obs, byrow = TRUE)
mean_distribution <- colMeans(simulated_matrix)
# Imprimer la moyenne et l'ecart type estimés
mean(mean_distribution)
sd(mean_distribution)
# Faire le graphique de la distribution simulée
plot <- ggplot(data.frame(obs = mean_distribution), aes(obs)) +
geom_histogram(aes(y = ..density.., color = "Simulated"),
bins = 30) +
stat_function(fun=function(x)dnorm(x, mean = args$mean, sd = args$sd),
size = 2, aes(color = "True")) +
scale_y_continuous("Density") +
scale_x_continuous("x") +
scale_colour_manual("Distribution", values = c("gray", "red")) +
theme_classic()
# Sauvegarder le graphique
if (args$graph_save) {
ggsave(args$path_graph, plot)
}Voici un exemple d’appel (voir figure) et de résultat effectués à la ligne de commande en utilisant ce programme :
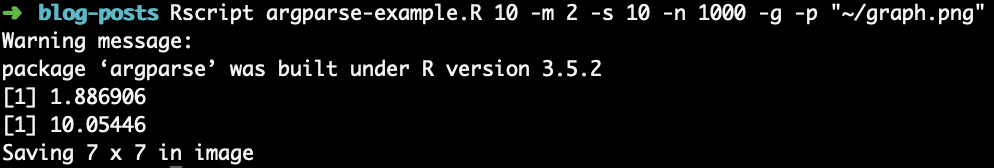
Figure 1: Exemple d’appel du programme à partir de la ligne de commande.
Conclusion
Pour davantage d’informations sur le fonctionnement de argparse et des différentes options
qu’il offre, vous pouvez consulter le dépôt du
paquetage et aussi la documentation complète
en ligne de la même librairie Python.
J’espère que ce court article vous aura permis de comprendre l’essentiel quant au fonctionnement des
interfaces de ligne de commande en R et leur utilité.





